 Федеральное государственное бюджетное учреждение науки
Федеральное государственное бюджетное учреждение наукиИНСТИТУТ БИОЛОГИИ РАЗВИТИЯ им. Н.К. Кольцова РАН
Koltzov Institute of Developmental Biology of Russian Academy of Sciences

|
|
Лаборатория БИОИНФОРМАТИКИ И МОЛЕКУЛЯРНОЙ ГЕНЕТИКИКоллектив лаборатории
Основные направления исследований:Лаборатория создана в 2022 году в рамках реализации программы Министерства образования и науки РФ № 075-15-2021-1075 «Генетические технологии». Основным направлением исследований, проводимых в лаборатории, является изучение молекулярной и клеточной организации мозга и сенсорных систем в контексте моделей нарушения развития нервной системы, метаболических, эндокринных и психиатрических заболеваний и нейродегенеративных расстройств. Базовый применяемый экспериментальный подход - секвенирование РНК одиночных клеток (scRNA-seq) интересующего отдела мозга. Ключевым преимуществом данного подхода является возможность изучать активность генов в каждой отдельной клетке в образце, что позволяет получить понимание о генной активности в принципиально ином разрешении. Основным этапом работы является анализ данных секвенирования. В ходе анализа применяется весь широкий арсенал существующих вычислительных методов, которые были разработаны специально для анализа данного типа эксперимента. Помимо готовых решений, коллектив лаборатории занимается разработкой и оптимизацией алгоритмов и методов, которые позволяют решать задачи транскриптомики одиночных клеток.
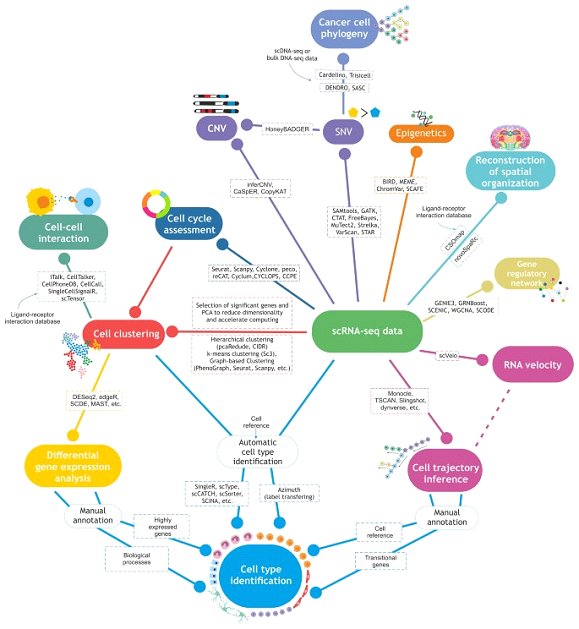
Существующие подходы анализа данных секвенирования РНК одиночных клеток (Khozyainova et al, Biochemistry (Moscow), 2023)
Модели диабета 1-го и 2-го типа В настоящее время, диабет 1-го и 2-го типа является социально значимым заболеванием и серьезной проблемой: порядка 8.8% взрослого населения во всем мире классифицируются как страдающие диабетом и приводит к ряду тяжелых метаболических нарушений и ожирению (тип 2). Глобальный уровень проблемы бросает вызов общей системе здравоохранения в связи с высоким риском сопутствующих заболеваний. Ранее была показана значительная роль гипоталамуса в регуляции метаболических процессов при диабет-связанных нарушениях, в частности, в контроле веса и стабильной работе эндокринных желез. В качестве модели диабета используются стрептозотоцин-индуцированная модель диабета 1-го типа (150 мг/кг) и стрептозотоцин-индуцированная модель диабета 2-го типа (30 мг/кг инъекция в неонатальных животных). Объект исследования - клеточные фенотипы и их изменения в гипоталамусе - нейроэндокринном центре мозга. Анализ данных проводится на результатах секвенирования РНК одиночных клеток (scRNA-seq) из контрольной и экспериментальной групп животных, а также на доступных релевантных данных из опубликованных работ по теме исследования. 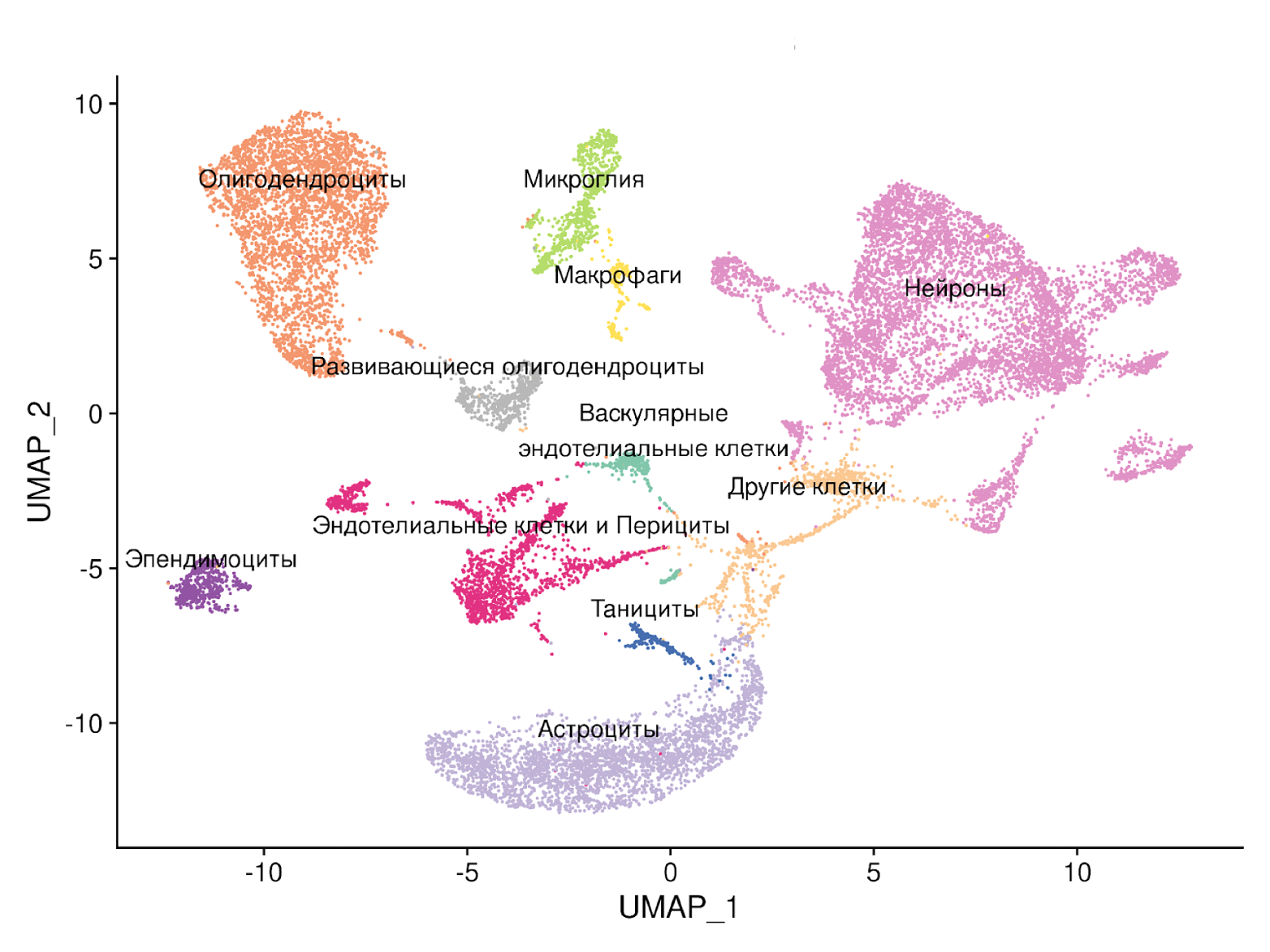
Распределение клеточных типов в наборе данных секвенирования одиночных клеток гипоталамуса в условиях диабета I типа при сравнении с контрольными образцами. 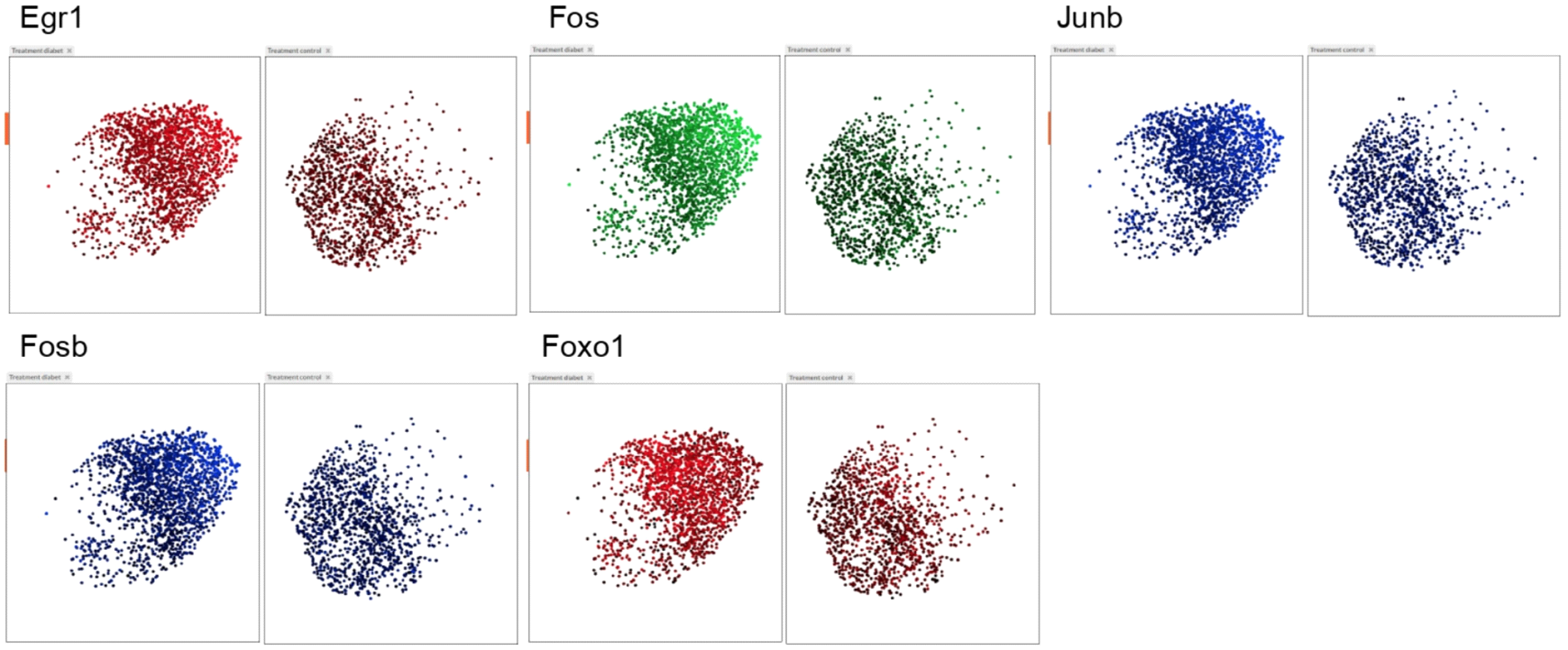
Распределение экспрессии генов регулон, наиболее специфичных для образцов диабета I типа. Графики построены в пространстве координат UMAP на основе AUC значений регулонов, где каждая точка обозначает проекцию определенной клетки, а интенсивность цвета меняется от минимальных к максимальным значениям уровней экспрессии. Для каждого регулона график разделен на две части: левая содержит только клетки, относящиеся к образцам диабета I типа, правая содержит только клетки контрольных образцов. Изображения получены в веб-сервисе SCope на основе loom файлов, генерируемых программой SCENIC. Транскриптомные изменения при обучении страху Миндалевидное тело относится к лимбической системе мозга и играет важную роль в формировании эмоций, особенно страха, и процессах, связанных с памятью и обучением. Перечисленные функции обусловливают важное значение амигдалы в формировании тревожных расстройств и существенную роль в патогенезе тревожности при ряде расстройств аутистического спектра, ухудшая качество жизни людей, особенно социальную интеграцию детей. Механизмы синаптической пластичности, индуцированные обучением страху, во многом известны и продолжают изучаться, однако в настоящее время нет полной характеристики транскриптома амигдалы на уровне одиночных клеток в моделях обучения страхом. Изучение транскрипционных изменений в ответ на стрессовые воздействия и индукцию страха у животных позволит определить значимые механизмы, свойственные для конкретных клеточных типов. 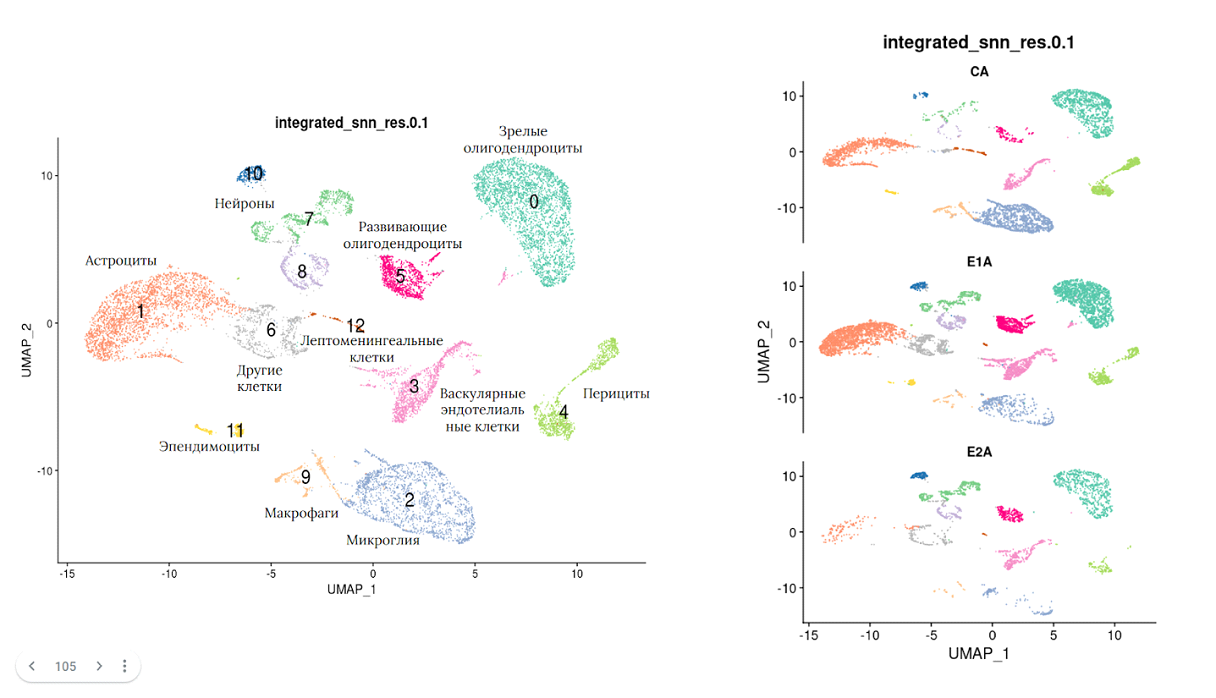
Распределение клеточных типов в наборе данных секвенирования одиночных клеток миндалевидного тела. График построен в пространстве координат UMAP, где каждая точка является проекцией одиночной клетки, подвергшейся секвенированию. 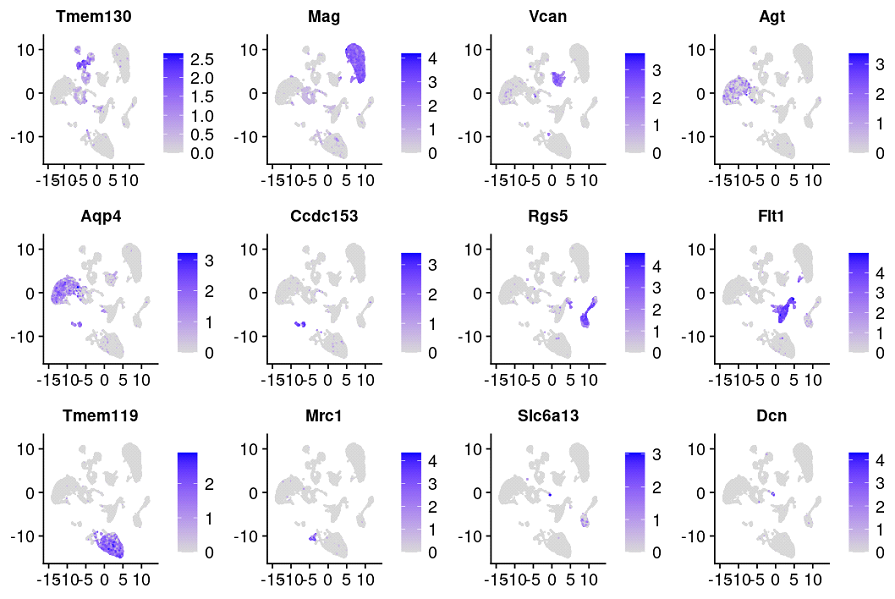
Распределение экспрессии маркерных генов относительно одиночных клеток. График построен в пространстве координат UMAP, где каждая точка является проекцией одиночной клетки, а интенсивность цвета показывает уровень экспрессии гена в клетке от минимальных значений (серый) до максимальных (синий). Избранные публикации:
|









