Комментарий к статье S. Deng et al. RNA m6A regulates transcription via DNA demethylation and chromatin accessibility. Nat Genet. 2022 Sep;54(9):1427-1437. doi: 10.1038/s41588-022-01173-1. Epub 2022 Sep 7.
 © Михайлов Виктор Сергеевич, главный научный сотрудник. Доктор биологических наук, профессор.
© Михайлов Виктор Сергеевич, главный научный сотрудник. Доктор биологических наук, профессор.Область научных интересов: репликация ДНК, репарация ДНК, ДНК-полимеразы, ДНК-связывающие белки, макромолекулярные синтезы в раннем развитии, репродукция вирусов.
Эпигенетическая информация кодируется химической модификацией азотистых оснований в ДНК и аминокислот в белках хроматина. В ДНК метилируется цитозин с образованием 5-метилцитозина (5mC), а в хроматине могут метилироваться лизины К36 и К9 в гистоне Н3 (H3K36me3, H3K9me2 и H3K9me3). Каким образом вносятся и удаляются эпигенетические модификации до недавнего времени оставалось неизвестным. В ряде недавних работ получены указания на то, что пусковым сигналом для модификаций в хроматине может являться метилирование аденозина в РНК. В рецензируемой работе показан элегантный механизм удаления 5mC из ДНК (деметилирования цитозина) после метилирования аденозина в новообразованной мРНК в ходе транскрипции гена РНК-полимеразой II. Эта статья явилась итогом десятилетнего исследования многочисленного коллектива ученых из Ракового центра Университета имени Сун Ят-сена в Гуанчжоу и других медицинских учреждений КНР. Первые парные образцы злокачественных и здоровых тканей от больных плоскоклеточной карциномой пищевода (ESCC) для исследования были получены ими в 2012 г. Помимо клеточных линий ESCC человека KYSE30 и KYSE510 в работе использовали клетки эмбриональной почки человека 293Т (НЕК293Т, SCSP-502).
Согласно предложенной авторами и экспериментально доказанной ими модели удаление 5mC осуществляется согласованной активностью трех белков: метилтрансферазы METTL3, белка FXR1 (m6A reeder), узнающего m6A в РНК, и ДНК-деметилазы TET1 (Рис. 1).
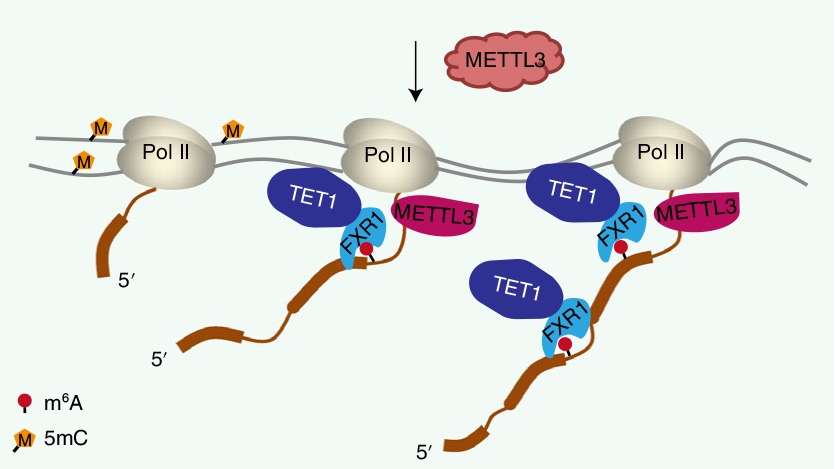
Процесс удаления 5mC запускается метилированием ферментом METTL3 аденозина в новосинтезированной мРНК после считывания РНК-полимеразой II участка ДНК-матрицы с 5mC. Почему фермент метилирует аденозин в мРНК после прочтения определенного 5mC в матрице и не делает этого после прочтения других матричных 5mC остается неясным. Возможно, процесс метилирования m6A в мРНК носит стохастический характер, либо метилирование регулируется контекстом соответствующих нуклеотидных мотивов. При этом, модификации ближайших гистонов на образование m6A в мРНК не влияют. После синтеза m6A в мРНК в дальнейших реакциях удаления 5mC из ДНК метилтрансфераза METTL3 не участвует. Новообразованный m6A эффективно узнается и быстро связывается белком-сканером FXR1, который находится в функциональном взаимодействии с деметилазой TET1 и активирует этот фермент в отношении соответствующего 5mC в матричной ДНК. Деметилаза TET1 тесно прилегает к РНК-полимеразе II и вероятно взаимодействует с ней (Рис. 2). Удаление 5mC из ДНК сопряжено с процессом транскрипции.
Интересно, что FXR1 и ТЕТ1 не имеют специфических сайтов для узнавания друг друга. Их функциональное взаимодействие обусловлено формированием белком FXR1конденсата жидкость-жидкость (liquid-liquid phase separation) с участием структурно дезорганизованного домена белка. Фазовое разделение является малоизученной, но широко распространенной формой формирования агрегатов функционально связанных белков, которые физически не связаны друг с другом и не изолированы от других компонентов клетки мембраной. Недавно показано, что фазовое разделение играет важную роль в формировании репликативного аппарата клетки, образовании комплексов патогенных белков в ядрах клеток нервной системы, изоляции убиквитинированных белков в цитоплазме при стрессе и целом ряде других процессов. Разделение фаз является, по-видимому, универсальным способом функциональной компартментализации в клетке – феномена известного довольно давно. В эксперименте образование комплексов белков за счет разделения фаз регистрируется по формированию «капель», в которых белки утрачивают способность к независимому друг от друга перемещению внутри клетки.
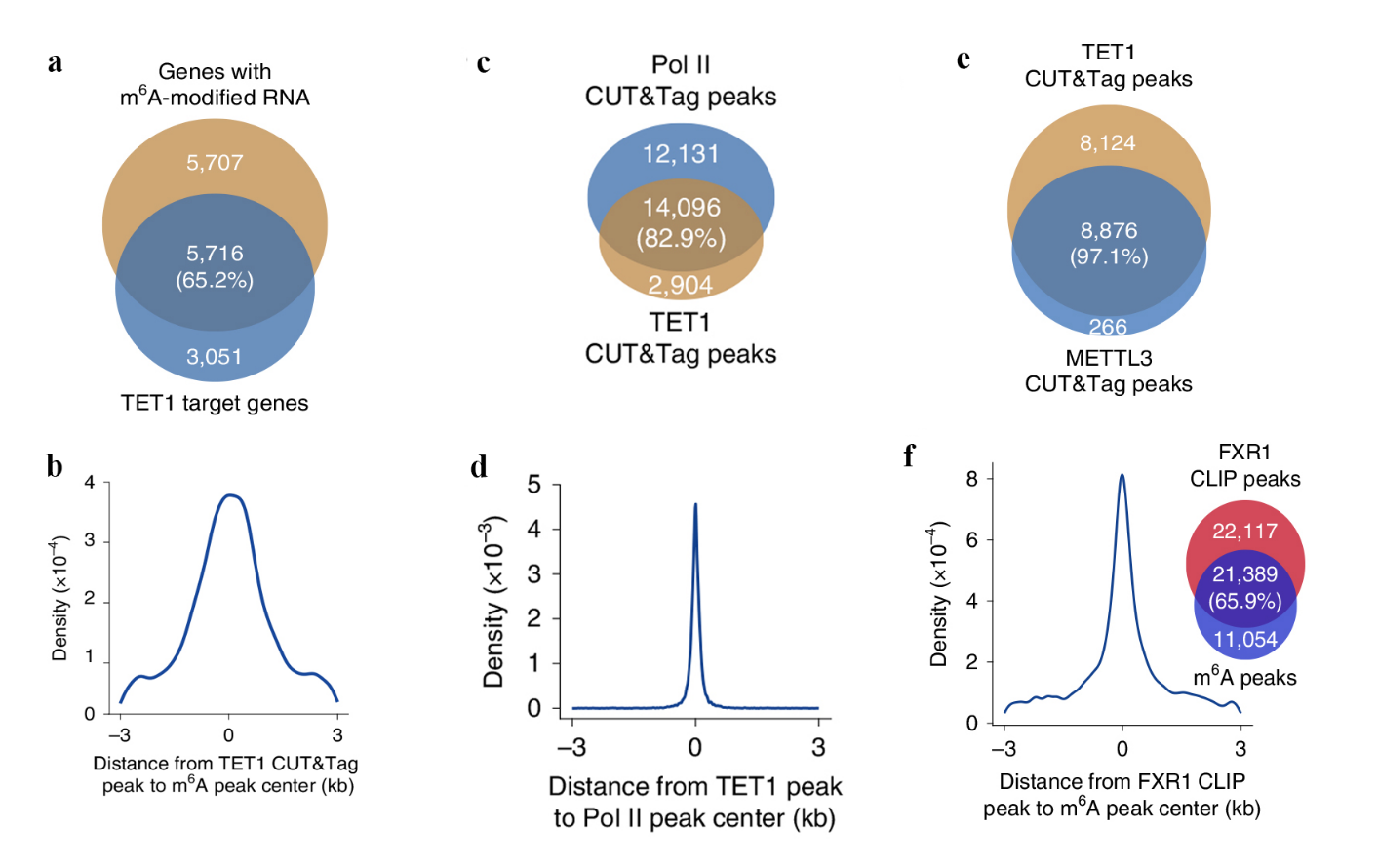
Основным аргументом того, что все три компонента METTL3, FXR1 и TET1выполняют свои роли в составе единого функционального комплекса является их ко-локализация на хроматине в местах, обогащенных m6A в РНК (Рис. 2), а также обогащенных 5mC и промежуточными продуктами деметилирования 5mC островками CpG в ДНК. Для получения этих результатов были использованы разнообразные современные методы геномного и функционального анализа. Распределение 5mC и m6A в хроматине изучали методом фермент-связанной иммуносорбции и иммуноферментного анализа (ELISA) в сочетании с масс-спектрометрией LC-MS/MS. Проводили параллельное m6A секвенирование РНК (MeRIP-sequencing) и полногеномное секвенирование. Распределение белков изучали с помощью секвенирования после иммунопреципитации хроматина (ChIP-seq). Использовали различные методы иммунопреципитации хроматина в сочетании с Вестерн-блоттингом и масс-спектрометрией LC-MS/MS. Функция белков METTL3, FXR1 и TET1 в удалении 5mC была изучена в опытах с мутантными формами ферментов, а также с помощью нокаутов соответствующих генов.
Важным источником информации в ходе исследования служили также публично доступные биоинформатические базы данных. Их широкое использование на протяжении всего проекта является отличительной характеристикой рецензируемого исследования. Так, экспериментальную работу предварял тщательный анализ публичных баз данных на наличие указаний о взаимосвязи m6A в РНК и 5mC в ДНК. Среди использованных в работе биоинформатических источников были базы для эмбриональных стволовых клеток мыши (mESC), база KEGG была использована для функциональной аннотации генов с измененным уровнем метилирования m6A в РНК и 5mC в ДНК. Собственные оригинальные результаты авторов были также выложены в общедоступную базу данных (GEO accession GSE168344).
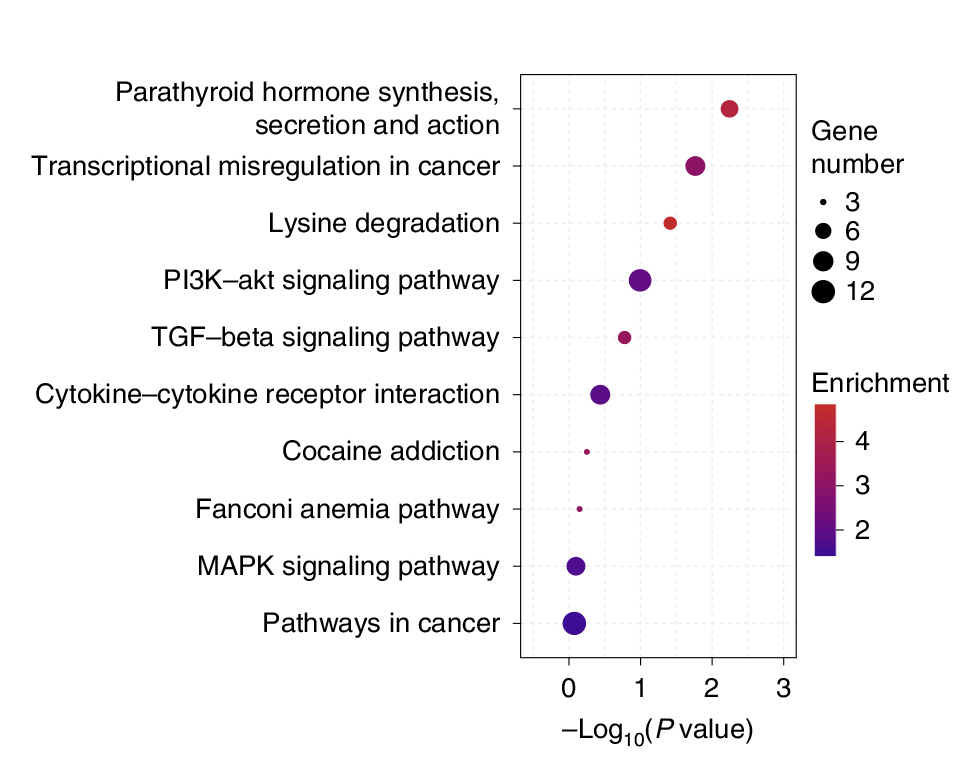
Активация процесса деметилирования ДНК в результате взаимодействия m6A РНК и 5mC ДНК сопряжена с ремоделированием хроматина и изменением уровня транскрипции генов. Об этом свидетельствует изменение степени доступности (открытости) хроматина. Декомпактизация хроматина обычно хорошо коррелирует с увеличением уровня транскрипции генов. Клетки плоскоклеточной карциномы пищевода (ESCC), которые изучали в ходе данной работы, характеризовались более высоким уровнем m6A и более высоким уровнем экспрессии ряда генов по сравнению с контрольными клетками из нормальных тканей. При этом уровень экспрессии ключевых факторов METTL3, FXR1 и TET1 в образцах ESCC был существенно выше, чем в прилегающих нормальных тканях. Таким образом, процесс деметилирования ДНК, сопряженный с индукцией m6A, может существенно нарушать регуляцию транскрипции в тканях и с большой вероятностью способствовать их злокачественной трансформации. Аннотация генов с гиперметилированной m6A РНК показала высокий уровень их ассоциации с нарушением регуляции транскрипции в злокачественных клетках (Рис. 3). Таким образом, факторы METTL3, FXR1 и TET1 могут выступать потенциальными терапевтическими мишенями для лечения онкологических заболеваний.
В научной литературе в последние годы появляются свидетельства того, что m6A метилирование РНК может также регулировать процесс эпигенетической модификации гистонов в хроматине. Таким образом, метилирование РНК может выступать в качестве универсального способа эпигенетической регуляции экспрессии генов. Ссылки на эти работы можно найти в кратком сообщении
Wenqi Xu and Hongjie Shen, When RNA methylation meets DNA methylation. Nat Genet. 2022 Sep;54(9):1261-1262. doi: 10.1038/s41588-022-01166-0. Epub 2022 Sep 7.
Новость подготовил © Михайлов В.С.
25.10.2022