 © Куликов Алексей Михайлович,
зав. лабораторией эволюционной генетики развития. Д.б.н., зам. директора ИБР по научной работе.
© Куликов Алексей Михайлович,
зав. лабораторией эволюционной генетики развития. Д.б.н., зам. директора ИБР по научной работе.Область научных интересов: механизмы эволюции на ранних этапах дивергенции видов.
С-парадокс (David Comings и Susumu Ohno) свидетельствует об огромной избыточности генома, в котором островки кодирующих последовательностей погружены в море некодирующей ДНК. В состав некодирующей ДНК входит множество функционально-активных последовательностей, от мобильных элементов и разнообразных некодирующих РНК до структурно и регуляторно-значимых последовательностей. Реализация генетической информации зависит от согласованной работы большинства этих последовательностей. Можно ожидать, что функциональная активность генов, и особенно консервативных генов домашнего хозяйства, поддерживается стабильным паттерном регуляторных элементов. Этот паттерн должен наследоваться видами потомками от общего предка и постепенно меняться в ходе эволюции. Но это не так.
Изменчивость некодирующих последовательностей ДНК представляет собой интересную эволюционную загадку, демонстрируя кардинальные изменения области промотора и расположенного выше межгенного спейсера даже у таких консервативных генов, как ras85D дрозофилы. Представляя собой молекулярный переключатель, кодируемый этим геном белок сохраняет структуру доменов и функциональных сайтов на протяжении эволюционной истории многоклеточных животных. Его молекулярные и биологические функции остаются неизменными у разных видов дрозофил. В то же время изменчивость области промотора, межгенного спейсера и значительной части 5’UTR свидетельствуют о независимом эволюционном происхождении этих последовательностей. Как часто и каким образом происходила замена этих участков, каким образом могла при этом сохраняться и поддерживаться функциональная активность гена?
Для ответа на эти вопросы были исследованы соответствующие последовательности 38 видов дрозофил, определены эволюционно-консервативные мотивы (ECM) в составе некодирующих последовательностей. Распределение ECM позволило уточнить границы области, имеющей общее эволюционное происхождение у исследованных видов. В составе разных подродов и групп видов эти границы имеют отличающуюся локализацию и смещаются у подрода Drosophila вплоть до первого интрона, разрывающего область 5’-некодирующей последовательности примерно посередине. Очевидно, в ходе эволюционных перестроек этой области происходила неоднократная смена промотора и расположенных ниже последовательностей. Особенный интерес вызывает ECM4 (рис.1), помеченный фиолетовым цветом и «прижатый» к области промотора справа. У видов дрозофил из подродов Drosophila и Sophophora он расположен с небольшим смещением относительно промотора, но самое главное, на противоположных цепях хромосомы, в составе участка последовательности, имеющей независимое эволюционное происхождение у обоих подродов дрозофил, что предполагает механизм конверсионного восстановления функционально-значимой последовательности сразу после события перестройки данной области в одной из эволюционных линий.
Последовательное изменение области некодирующей последовательности в разных эволюционных линиях изученных 38 видов дрозофил было сопряжено как минимум с 26-ю инсерционными событиями, в том числе 18-ю в районе между генами, тремя непосредственно в область промотора, четырьмя в левой половине 5’UTR и одной инсерцией в первый интрон. События встраивания и делеции или деградации транспозонов в область промотора ras85D в ходе эволюционной дивергенции видов дрозофил картированы на филогенетическом дереве видов (рис.2). Участие в таком преобразовании последовательности в разных эволюционных линиях приняли транспозоны всех известных классов – от ДНК-транспозонов до ретротранспозонов с длинными концевыми повторами и без них. Сохраняющаяся гомология привнесенного инсерцией фрагмента у родственных видов свидетельствует, что удаление избыточной последовательности в отдельных эволюционных линиях происходит фрагментарно, с разной скоростью в разных линиях. Более поздние инсерции выявляются на выравнивании как вставки значительной длины, с выраженной гомологией к тому или иному транспозону по краям вставки. В двух случаях транспозиции привели к внутрихромосомной неаллельной рекомбинации между гомологичными копиями транспозонов и формированию инверсионных перестроек, также полностью изменивших структуру межгенного района и значительной части 5’UTR ras85D у D. willistoni и у общего предка дрозофил группы virilis. Цепь рекомбинационных событий, сопровождавшаяся последовательными инверсиями, сформировавшими современный порядок генов у видов группы virilis, реконструирована на схеме 3. Сохранившиеся фрагменты инвертированных повторов от рекомбинировавших копий древнего ДНК-транспозона, накопившие после события рекомбинации независимую эволюционную изменчивость, позволяют оценить время формирования первичной перестройки у предкового вида порядка 20 млн. лет, вскоре после дивергенции предковых видов групп repleta и virilis.
Глобальные перестройки некодирующей области гена ras85D сопровождались сменой структуры промотора, состав элементов которого демонстрирует три основных паттерна (рис.4). Во всех случаях основная экспрессия гена осуществляется с широкого промотора или “широкого с пиком”. Значительная часть последовательности промотора (54 нуклеотида) у видов групп montium, obscura и ananassae, демонстрируют выраженную гомологию не только между собой, но также с представителем рода ScaptoDrosophila – S. lebanonensis, что указывает на предковое состояние данного участка у этих трех групп видов. Для области промотора у данных видов характерно наличие элементов CCAAT-box, TATA-box, Initiator (Inr), Downstream Promoter Element (DPE), Ohler1 и Ohler7, и значимое обогащение по последним двум элементам (вариант 1 на рис. 4). Участок ограниченной гомологии в области промотора у D. willistoni с последовательностью промотора указанных выше видов включает элементы BRE down (BREd), Ohler1, Inr, CCAAT-box (вариант 1а на рис. 4). Область одиночных стартов у D. willistoni находится на 200 п.о. выше основного промотора. Ей соответствует локализация элемента Ohler7, в паре с элементом DRE, и каноническое положение элементов TCT и Inr-DPE.
Большинство видов, относящихся к группе melanogaster подрода Sophophora, демонстрирует обязательное присутствие элементов Ohler7 и DRE (вариант 2 на рис.4). В этой области также встречаются элементы BREd, Inr и DPE, обогащение которыми не подтверждается достоверно относительно последовательности межгенного района, и распределенные случайным образом внутри самого промотора. У видов D. eugracilis и D. suzukii из группы melanogaster качественные изменения последовательности межгенного спейсера захватывают область промотора, характерного для других видов этой группы, частично у D. eugracilis и полностью у D. suzukii (варианты 2а и 2b’ на рис.4). У D. eugracilis делетирована большая часть спейсера, а область промотора осталась в том же положении относительно ECM4, но утеряла элемент DRE и приобрела элементы TATA-box и Ohler1. У D. suzukii точка старта транскрипции TSS сместилась выше на 100 п.о., в соответствии с данными SRA. Она подтверждается согласно данным по позициям ридов из библиотеки TSA, и добавляется еще одна, расположенная дистальнее на 250 п.о. В обеих TSS 5’-концы дистальных ридов расположены левее элементов Ohler1. Во второй, наиболее удаленной TSS обнаруживаются также элементы TCT и BREd, в ближней – дополнительно Inr с TATA и BREd в каноническом положении, и расположенные downstream Ohler6, Ohler7, Ohler10, и на комплементарной цепи – DRE.
Виды подрода Drosophila проявляют выраженную гомологию и уникальный для этих видов паттерн области промотора (вариант 3 на рис.4). Она включает ECM7, расположенный выше фрагмент длиной около 40 п.о. и половину расположенного на комплементарной цепи ECM4. Состав элементов промотора значимо обогащен мотивами Ohler6 и Ohler7. Элемент DRE не показывает значительного обогащения относительно последовательности всего спейсера, но его положение в определенной позиции промотора неслучайно. Элемент Ohler7 в этом промоторе всегда расположен перед последовательностью ECM7 на «-»-цепи, элементы DRE расположены: первый – перед элементом Ohler7, и второй – в начале ECM4. Элемент Ohler6 также встречается преимущественно на «-»-цепи. Последовательность промотора обогащена коровыми элементами INR, DPE, CCAAT-box и BREd, относительно их встречаемости в составе межгенного спейсера, но их неслучайное распределение внутри самой последовательности промотора не подтверждается. Пик экспрессии в составе широкого промотора ассоциирован с элементами Ohler6 и Ohler10.
Bид D. busckii подрода Drоsilopha не имеет сколько-нибудь заметной гомологии c остальными видами в последовательности, маркированной TSS. Тем не менее, размер 5’UTR транскриптов примерно соответствует таковым остальных видов, и в области старта присутствуют элементы Ohler1, CCAAT-box, Ohler6, Ohler7 (на противоположной цепи) и ТАТА-box.
Состав родственных структур промоторов, представленных у каждого из трех наборов видов, может меняться в связи с накопившимися точечными заменами, инсерциями/делециями и граничащими с промотором перестройками, сохраняя, тем не менее, свои основные черты, особенно в правой, базальной части промотора. Все выявленные структуры несут в качестве основных, постоянно присутствующих мотивов Ohler elements, чаще всего Ohler1, Ohler6 и Ohler7, причем два последних элемента обнаруживаются также на противоположной цепи.
Общая картина изменчивости всей некодирующей последовательности позволяет предположить, что основную роль в регуляции экспрессии гена ras85D играют мотивы, расположенные в области 5’UTR и 1-го интрона. Интерес представляют восемь мотивов, выявляемых во всех случаях вне зависимости от примененных вариантов анализа, проявляющих структурное постоянство, имеющих постоянные или в значительной степени перекрывающиеся позиции в составе анализируемой последовательности. На рис. 1 это мотивы ECM1, ECM2, ECM3, ECM4, ECM5, ECM7, ECM12 и ECM13. Был проведен анализ обогащения данных мотивов потенциальными сайтами связывания транскрипционных факторов дрозофилы. Результаты анализа были отфильтрованы по известным для D. melanogaster 43-м транскрипионным факторам (TF) и их партнерам по транскрипционному комплексу из 20 суперсемейств TF и четырех семейств белков коактиваторов и корепрессоров. Статистическая оценка обогащения восьми выбранных ECM сайтами связывания TF показала, что 23 из них неслучайным образом распределены по этим последовательностям. Наиболее часто представлены сайты связывания TF lola и kni – в составе шести и четырех ECM соответственно. Обогащение сайтами связывания для TF D, disco, Dll, en, Med, pnr, Ubx показано в составе 3х ECM и для TF – bab1, BEAF-32, sens, Trl, twi показано в составе двух ECM. GO анализ обогащения биологических процессов, проведенный по спискам транскрипционных факторов, специфичных для каждого мотива, показал функциональные различия ECMs. Так, действие ECM2 на экспрессию гена ras85D опосредует его участие в контроле деления клетки, морфогенезе слюнных желез, системы трахей, иннервации сетчатки, сегментации эмбриона и клеточных ответах на BMP и экдизоновые стимулы. ECM1 необходим для контроля развития ножных, крыловых, глазных и генитального дисков и передне-задней поляризации клеток. ECM3 и ECM12 опосредуют участие гена в дорзо-вентральной поляризации клеток, развитии нервной системы, дифференциации нейронов и фоторецепторов, формировании кутикулы; ECM5 – в сегментации эмбриона и развитии сердца, циркуляторной системы, лимфатической железы и периферической нервной системы; ECM7 – в регуляции митотического цикла, формировании нейробласта и дифференцировке стволовых клеток, дифференцировке хет и развитии периферической нервной системы и мальпигиевых сосудов.
Можно заключить, что высокая изменчивость препромоторной области и дистальной части 5'UTR связана с перестройками, вызванными вставками мобильных элементов. Эти части последовательности имеют независимое эволюционное происхождение. Критические для регуляции процессов развития и функционирования клеток мотивы (ECM) располагаются ниже промотора и вместе с промоторной областью могут быть перенесены в эволюционно новые последовательности с помощью механизмов генетической конверсии. Эволюционные изменения в регуляторной области консервативного гена, приводящие в большинстве случаев к его инактивации или аномалиям, несовместимым с нормальным развитием, могут быть восстановлены до приемлемого уровня в кратчайшие сроки, практически мгновенно в масштабе эволюционного времени. Наконец, отметим еще один неожиданный вывод. Исходя из общих представлений о формировании механизмов адаптации и изолирующих барьеров в ходе эволюции, консервативные гены должны поддерживать гомеостаз организма и служить фундаментом, на котором строятся все эволюционные инновации. Однако полученные результаты позволяют предположить, что периодические (но не критические) изменения регуляторной активности консервативного гена в ходе эволюции его некодирующей последовательности могут оказывать свое влияние на онтогенетический процесс, также приводя к эволюционно значимым изменениям.

Рис. 1. Полиморфизм межгенной области, промотора, 5'UTR и интрона 1 у видов дрозофил с разной степенью родства.
Схема распределения эволюционно консервативных последовательностей и инсерционно-делеционного полиморфизма в анализируемой области гена ras85D. Структура эволюционно консервативных последовательностей, полученных с помощью алгоритма MEME. На схеме прямоугольники над линией показывают расположение ECM на плюсовой цепи, а прямоугольники под линией показывают расположение ECM на минус-цепи. Уменьшенная высота прямоугольника свидетельствует о вырождении ECM. В верхней части представлены виды подрода Drosophila (D. virilis — D. albomicans), а в нижней — виды подрода Sophophora (D. willistoni — D. melanogaster). D. busckii (подрод Dorsilopha) располагается между подродами Drosophila и Sophophora.
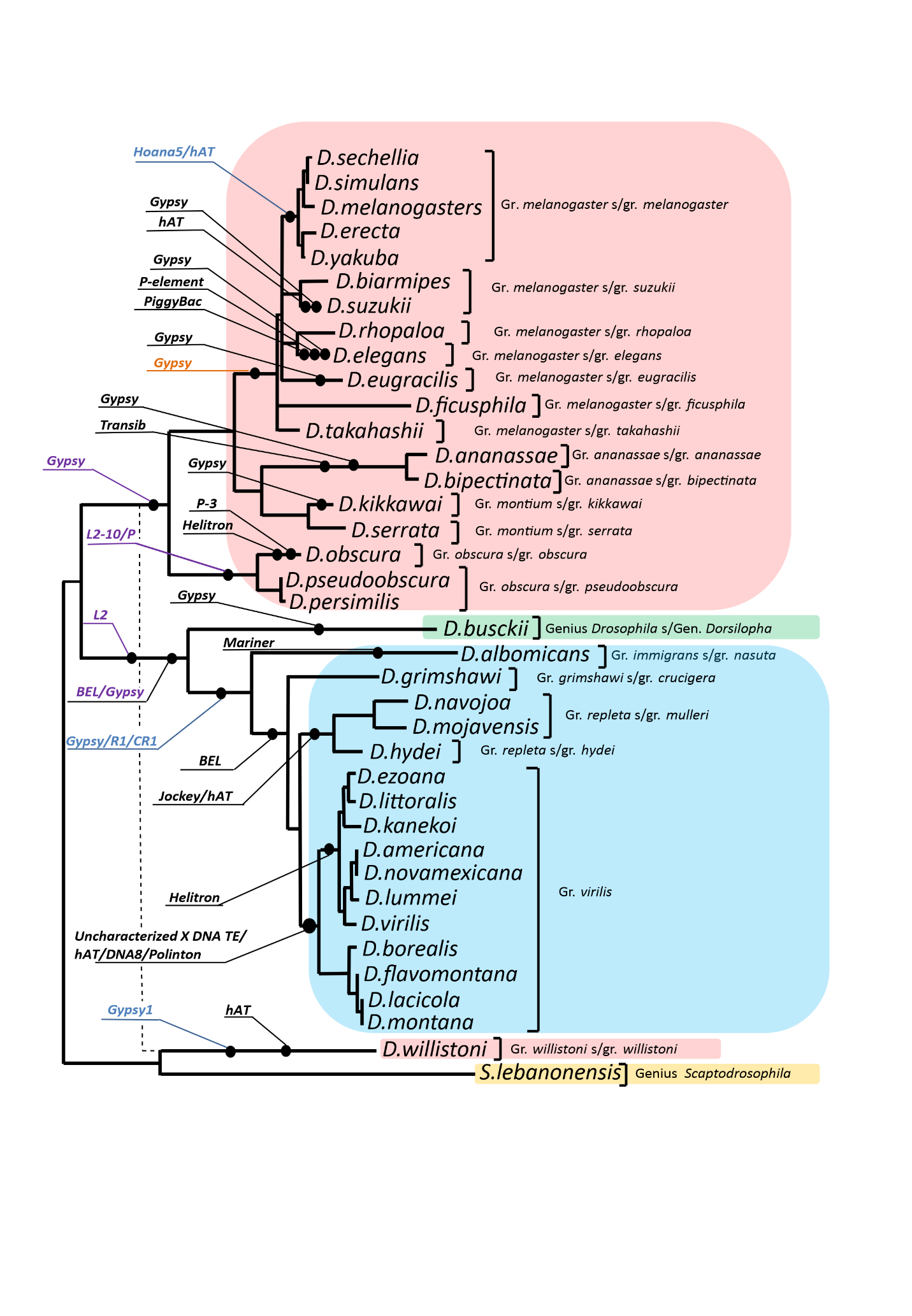
Рис. 2. Распределение идентифицированных событий вставки транспозонов на эволюционном древе анализируемых видов дрозофилы.
Филогенетическое дерево построено методом максимального правдоподобия на основе трехпараметрической модели Тамуры. Были использованы последовательности экзонов и 3'UTR гена ras85D 37 видов дрозофилы. Гамма-распределение использовалось для моделирования различий скорости эволюции между сайтами [5 категорий (+G, параметр 0,8107)]. Модель изменения скорости позволила некоторым сайтам оставаться эволюционно неизменными [(+I), 19,28% сайтов]. Структура полученного филогенетического дерева в целом согласуется с деревом видов, полученным для 155 геномов видов Drosophila (Suvorov et al., 2021), за исключением D. willistoni, положение которой на дереве видов указано пунктирной линией. Выноски определяют мобильный элемент и события вставки на ветвях эволюционного дерева. Обозначенные выноской вставки в следующих областях: межгенная область, промотор, 5'UTR и интрон 1 были выделены черным, синим, фиолетовым и оранжевым цветом соответственно.
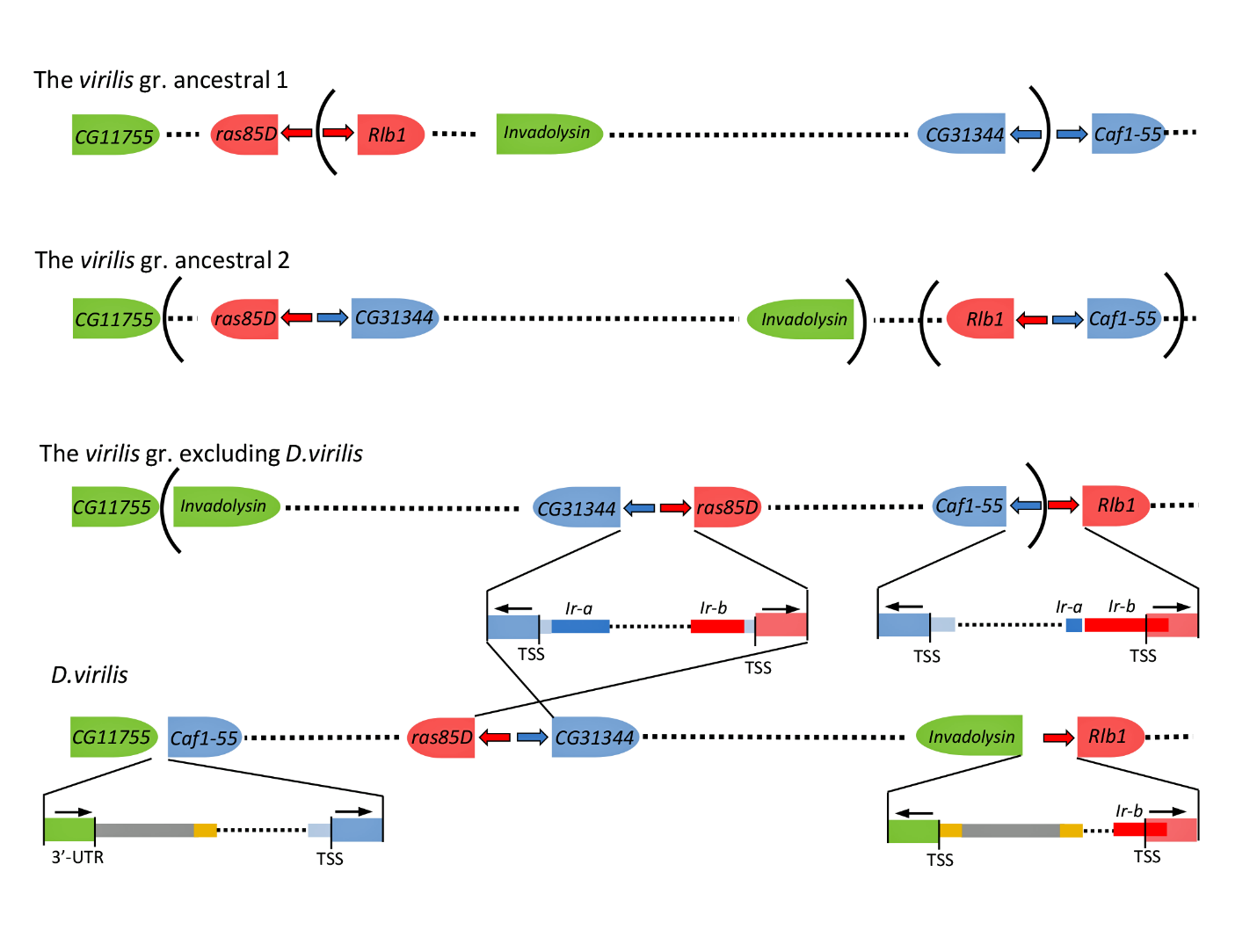
Рис. 3. Минимальное количество реаранжировок, необходимых для трансформации предкового состояния хромосомы 2 в специфичное для группы virilis.
Синие и красные стрелки — перевернутые повторы Ir-a и Ir-b, соответственно, которые отмечают места вставки двух копий древнего ДНК-X-транспозона в межгенные области ras85D—Rlb1 и CG31344—Caf1-55 у предкового вида группы virilis. Элементы DAIBAM окрашены в желтый цвет (перевернутые терминальные последовательности) и серый цвет (Reis, et al., 2018). Остатки наследственных последовательностей окрашены в голубой цвет. Номенклатура генов соответствует названиям ортологов D. melanogaster. Представление не в масштабе.
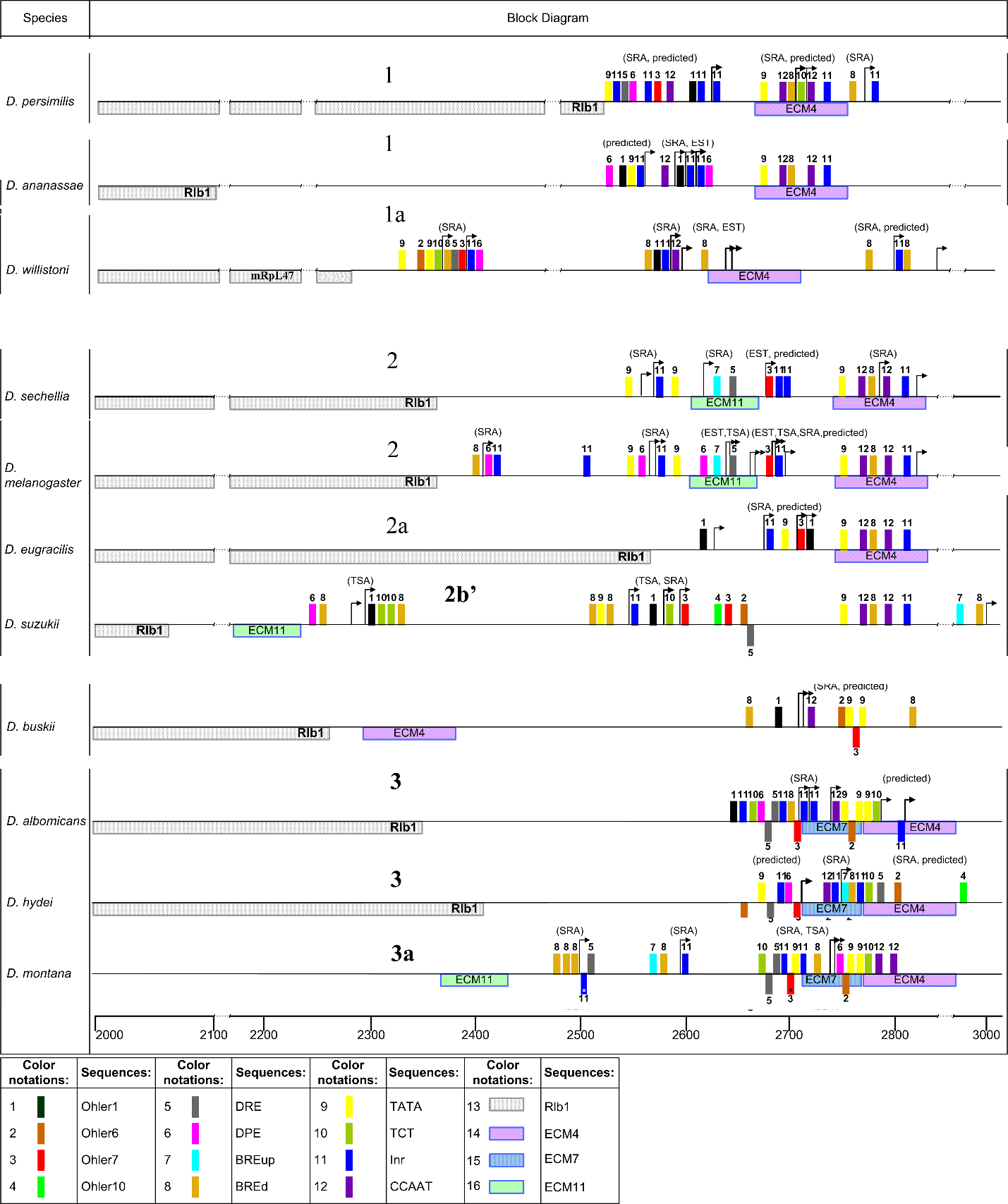
Рис. 4. Паттерны промоторных элементов в участках TSS гена ras85D и его ортологов у изученных видов дрозофил.
Шкала под линией показывает положение от начала общего выравнивания. Цветные прямоугольники представляют элементы промотора. Элементы, показанные под прямой линией, указывают на гомологию с минус-цепью ДНК. ТСС отмечены стрелками. Ген, расположенный выше по течению, отмечен в левой части диаграмм.
Новость подготовил © 21.01.2022 Куликов А.М.