
Статья Jain S. и соавторов в журнале Nature Сell Biology посвящена программе создания биомолекулярного атласа человека (Human BioMolecular Atlas Program, HuBMAP, https://portal.hubmapconsortium.org/). Программа направлена на создание трёхмерного биомолекулярного атласа человеческого организма с разрешением в одну клетку.
Программа основана в 2018 году, и первый её этап (2018-2022) был посвящён стандартизации и унификации методологии и номенклатуры, созданию цифровой инфраструктуры проекта. Учитывая масштабность проекта на фоне потребности биологической науки в единстве методологии и номенклатуры, принятые в HuBMAP стандарты могут послужить ядром интеграции.
Унифицированная терминология представлена в разработанном «Справочном атласе анатомии человека» (Human Reference Atlas, HRA, https://humanatlas.io/) и справочных таблицах (https://humanatlas.io/asctb-tables).
Опубликовано более 200 стандартизированных протоколов: https://www.protocols.io/workspaces/human-biomolecular-atlas-program-hubmap-method-development, https://humanatlas.io/standard-operating-procedures Контакты лиц, ответственных за соответствие экспериментальных протоколов эталону, приведены в соответствующих разделах по ссылке: https://software.docs.hubmapconsortium.org/assays.html
Среди методологических рекомендаций отдельно следует отметить разработку оптимизированных мультиплексных иммунофлуоресцентных панелей (ОМИП), предназначенных для пространственного картирования анатомических структур в 2D и 3D. Наличие рекомендованных ОМИП позволяет не только повысить воспроизводимость, но и избежать значительных затрат времени и средств, сопряжённых с разработкой таких панелей де ново. Разработанные стандартизированные панели антител для картирования органов представлены по ссылке: https://humanatlas.io/omap. *
*В настоящий момент (на 13.09.23) панели доступны для: лимфатического узла, кишечника, почки, кожи, печени, поджелудочной железы, лёгкого, плаценты, нёбной миндалины, селезёнки, сетчатки глаза.
С осени 2022 года стартовал этап набора экспериментальных данных для HuBMAP. Основное внимание уделяется масштабированию охвата и гетерогенности биологических переменных (разные возраста, половая и этническая принадлежность). Данные размещаются в открытом доступе на портале проекта: https://portal.hubmapconsortium.org/ (рис.1), наборы данных включают 2D и 3D анализы, детектирующие РНК, белки и метаболиты в масштабе от макромолекулярного и субклеточного (100 нм) уровня до уровня органа.
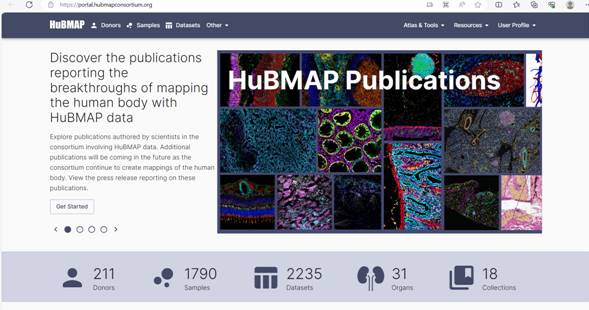
Рис. 1. Официальный сайт проекта; представлена статистика по размещённым на платформе материалам: на 13.09.23 – 211 доноров, 1790 уникальных образцов, 2235 биоинформатических наборов данных, 31 орган, 18 коллекций
При получении данных в рамках проекта используются: - аутофлуоресцентная микроскопия; - анализ доступного для транспозазы хроматина (ATACseq); - совместное обнаружение путём индексирования (CODEX); - визуализирующая массовая цитометрия (IMC); - послойная флуоресцентная микроскопия (light sheet fluorescence microscopy, lightsheeet); - жидкостная хроматография – масс-спектрометрия (LC-MS); - масс-спектрометрия с лазерной десорбцией / ионизацией с матрицей (MALDI-IMS); - масс-спектрометрия вторичных ионов (SIMS); - пространственная масс-спектрометрия; - раммановская спектроскопия; - оптическая когерентная томография; - мультиплексная иммунофлуоресценция (MxIF); - РНК-секвенирование (RNAseq), включая секвенирование единичных ядер/единичных клеток (sc/snRNA-seq), транскриптомику с пространственным разрешением; - секвенирование методом флуоресцентной гибридизации in situ (seqFISH); - полногеномное секвенирование (WGS); - пространственная молекулярная визуализация (CosMx); - др. (подробнее – https://software.docs.hubmapconsortium.org/assays.html). Сведение используемых методов в единую систему является одним из приоритетов проекта.
Все данные, полученные в HuBMAP, становятся общедоступными после тщательной оценки и контроля качества. В облачной базе данных хранятся необработанные результаты экспериментов, а также обработанные данные и опубликованные результаты последующего анализа. Доступны обширные метаданные, касающиеся биообразцов, анализов и протоколов (https://portal.hubmapconsortium.org, пользовательский интерфейс https://portal.hubmapconsortium.org/ccf-eui). Пользователи могут искать на портале HuBMAP доноров, образцы и наборы данных по полу, органу, типу данных и т. д. Результаты поиска отображаются в виде списка с дополнительными параметрами фильтрации и ссылками на экспериментальную, расчетную информацию и информацию о метаданных.
Кроме того, была выявлена функциональная асимметрия симметрично расположенного кластера CDC. Влияние ИЛ достоверно проявлялось в левом, но не правом кластере CDC. Эта различие может объяснятся несимметричным расположением нейронов мужской половой системы в ганглиях моллюска, который является гермафродитом. Эти нейроны расположены справа и оказывают тормозное влияние на «женский» кластер CDC.
В рамках проекта на сегодняшний день опубликовано более 250 статей, как методологических, описательных, так и аналитических, концептуальных. Подробнее с публикациями (на 13.09.23 – 252 статьи) можно ознакомиться по ссылке: https://commonfund.nih.gov/publications?pid=43
Перспективы программы сложно переоценить. Уже сегодня HuBMAP предоставляет огромный пул данных для анализа, составление же полного пространственного атласа радикально изменит биологическую науку.
Новость подготовила
© О.И. Сутягина
20.09.2023