

© Александр Финошин, аспирант лаборатории биохимии процессов онтогенеза. Область научных интересов: клеточная пластичность, протеомика, гомеостаз клеточного протеома, регенерация, металлопротеины.
© Ким Адамейко, младший научный сотрудник лаборатории биохимии процессов онтогенеза. Научные интересы: биоинформатика, транскриптомика, хроматин, беспозвоночные.
Junior research scientist Laboratory of Biochemistry. Interests: bioinformatics, transcriptomics, chromatin, invertebrates.
Благодаря новым методам на базе высокопроизводительного секвенирования и достижениям масс-спектрометрии мы узнаём всё больше о тонкой взаимосвязи различных уровней экспрессии генетической информации. Хорошо известно, что транскриптом клеток (совокупность мРНК) не определяет автоматически состав клеточного протеома (совокупности белков). Дифференциальная трансляции мРНК на рибосомах вносит существенный вклад в формирование протеома. Поэтому транслятом (совокупность мРНК, транслируемых на рибосомах в данный момент), является важным промежуточным этапом в реализации генетической информации. В статье Wang и соавторов, опубликованной в журнале Nature, впервые проведен сравнительный анализ транскриптомов, протеомов и транслятомов в клетках млекопитающих. Транслятом количественно характеризуют с помощью метода рибосомного профайлинга (Ribo-seq), основанного на изоляции защищённых рибосомами фрагментов мРНК от остальных видов клеточной РНК с последующим их секвенированием. Были получены данные RNA-seq и Ribo-seq для трёх органов (мозг, печень, семенники), формирующихся из разных зародышевых листков, пяти видов млекопитающих (человек, макака, мышь, опоссум и утконос) и курицы. Протеомные данные для человека и мыши взяли из предыдущего исследования. Анализ трёх уровней экспрессии генов у разных организмов позволил сделать интересные эволюционные выводы.
Транскрипция не всегда напрямую сопряжена с трансляцией [1]; анализ ранговой корреляции экспрессии на трёх уровнях (транскриптом, транслятом и протеом) подтвердил более тесную связь между «соседними» уровнями, чем между «крайними» - транскриптомом и протеомом (рис. 1A), что согласуется с данными о том, что эффективность синтеза белка является лучшим показателем содержания белка, чем мРНК [2]. При этом наиболее высокая корреляция наблюдалась между транскриптомным и транслятомным уровнями (0.78-0.90). Дисперсия экспрессии возрастала на 12-32% в транслятоме по сравнению с транскриптомом у всех организмов в соматических органах и падала в репродуктивном (рис. 1В), что говорит о тканеспецифичной трансляционной эффективности (ТЭ). Это наблюдается и на внутритканевом уровне. При анализе ТЭ в четырех типах клеток в семенниках мыши авторы установили, что кластер генов, избегающих общей трансляционной репрессии в сперматоцитах, действительно обогащен генами, связанными со сперматогенезом.
Исследуя ко-эволюцию экспрессионных уровней, авторы построили филогенетические деревья на основе экспрессии в транскриптоме и транслятоме клеток трёх тканей для примерно 5000 стабильно экспрессирующихся ортологов. В качестве попарного межвидового расстояния взята дисперсия изменений экспрессии (у близких видов она невелика), отнесённая к общей дисперсии экспрессии по всем видам. Таким образом, длины ветвей в деревьях отражают долю изменений экспрессии, связанную с эволюционными процессами. Интересно, что ветви транслятомных деревьев оказались на 20-22% короче, чем у транскриптомных (рис. 1С), что говорит о большей консервативности транслятомного уровня регуляции.
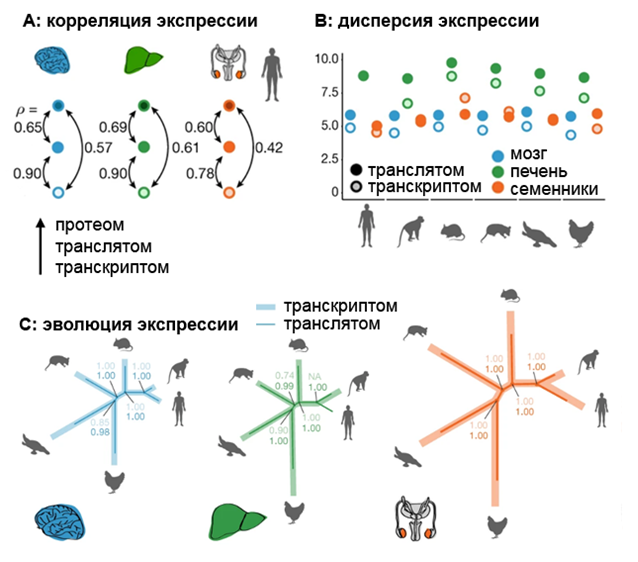
Рисунок 1. А – Ранговая корреляция Спирмана (Spearman’s p) между тремя уровнями экспрессии, рассчитанная по 9642 генам, стабильно экспрессирующихся в человеческих семенниках, мозге и печени. В – Дисперсия экспрессии на транкриптомном и транслятомном уровнях у шести видов в трех органах из разных зародышевых листков. C – Деревья, в которых длина ветвей соответствует доле дисперсии экспрессии, обусловленной эволюционными процессами.
Для интерпретации результатов авторы вводят понятие буферизации (“buffering”), или сглаживания изменений, происходящих на разных уровнях (примером могут служить нетранслирующиеся сплайс-изоформы, находящиеся под меньшим давлением отбора и вносящим вклад в большее разнообразие транскриптома). Они также исследуют скорости ко-эволюции экспрессии индивидуальных генов Δ, выводимые из межвидовых дисперсий. Генов, предположительно находящихся под стабилизирующим отбором (Δ<0, FDR < 0.1), оказалось в несколько раз больше, чем тех, которые находятся под положительным (Δ>0). В целом бо́льшая скорость эволюции в транскриптоме может ещё раз подтверждать бо́льшую гибкость регуляции этого уровня экспрессии генов.
Подробный анализ некоторых характеристик исследуемых ортологов показал, что гены, более чувствительные к мутациям, проявляют более низкую дивергенцию экспрессии на обоих уровнях вместе с более сильной буферизацией по сравнению с генами, которые относительно толерантны к мутациям (рис. 2). Эффект «важности» (essentiality) выше в соматических тканях по сравнению с семенниками и особенно силен в мозге. Для housekeeping генов (группа Broadly expressed) наблюдались очень низкие и в целом сходные скорости эволюции экспрессии. В отличие от housekeeping генов, тканеспецифичные гены эволюционируют с разной скоростью в разных органах. Гены, специфичные для мозга эволюционируют значительно медленнее, чем гены, специфичные для печени или семенников. Также установлено, что более эволюционно древние гены имеют более низкую дивергенцию экспрессии генов и более сильную буферизацию, чем у эволюционно молодых генов [6-7].
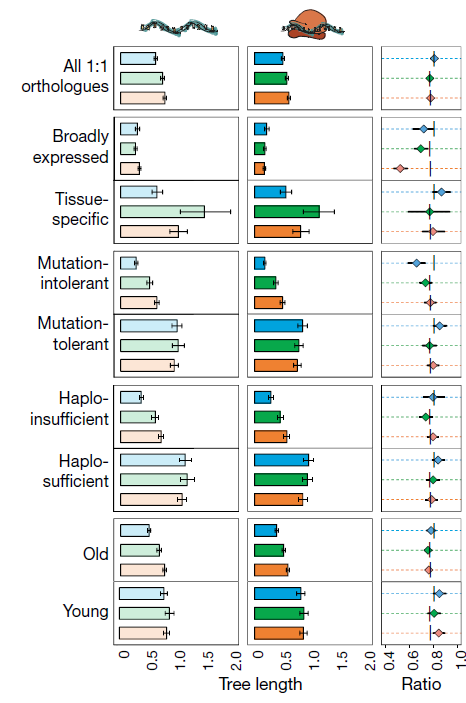
Рисунок 2. Экспрессионная дивергенция на двух экспрессионных уровнях была рассчитана для всех 8,109 стабильно экспрессирующихся ортологов (FPKM>1) для макаки, мыши и опоссума (референс) и для специфических групп генов: Broadly Expressed, Tissue specific, Mutation tolerant/intolerant, Haplo-insufcient/sufcient, old/young. Анализ был ограничен тремя видами, с целью увеличения количества 1:1 ортологов. Соотношения между скоростями дивергенции экспрессии генов (от трансляционной к транскрипционной) показаны справа от каждого набора гистограмм. Планки погрешностей рассчитанные на основе 1000 повторений (bootstrap).
Для понимания, в какой степени буферизация посредством активации трансляции могла ослабить эффект наличия лишь одной X хромосомы у самцов и/или эффект мейотического сайленсинга в половых хромосомах, сравнили экспрессионные уровни ортологов на современной Х хромосоме млекопитающих с более древними ортологами на “предковой” половой хромосоме (условная “предковая” половая хромосома была составлена из аутосомных ортологов курицы). Было установлено, что: 1. Эффективность трансляции для генов, сцепленных с Х-хромосомой плацентарных животных, значительно выше, чем для аутосомных генов, особенно в семенниках, тогда как для ортологов Х-сцепленных генов у яйцекладущих (курица и утконос) эффективность трансляции не выше по сравнению с другими аутосомными генами; 2. Более высокие уровни экспрессии X-сцепленных генов на транслятомном и протеомных уровнях по сравнению со транскриптомным уровнем наблюдаются во всех трех органах, с наиболее сильной активацией в семенниках; 3. Повышение уровня экспрессии от транслятома к протеому для X-сцепленных генов является относительно незначительным. Из этого следует, что повышенная экспрессия генов на X-хромосоме в основном обеспечивается транслятомным уровнем (например образованием полирибосом).
Авторы статьи предоставляют удобный веб-ресурс для изучения ко-эволюционных характеристик исследованных 5000 ортологов, который доступен по адресу https://ex2plorer.kaessmannlab.org/.
Полученные авторами результаты показывают важную роль транслятомного уровня регуляции экспрессии в реализации генетической информации и формировании клеточного протеома в разных органах эволюционно удаленных видов животных. Эволюционный процесс идет за счет более быстрого изменения тканеспецифичных генов и эволюционно молодых генов. При помощи веб-сервиса https://ex2plorer.kaessmannlab.org/ каждый может посмотреть данные по трансляционной эффективности интересующего гена, что, вероятно, поможет более точной интерпретации данных РНК-секвенирования. Результаты, полученные в данной статье, могут стать мощным стимулом в развитии и использовании метода рибосомного профайлинга.
1) Ingolia NT, Lareau LF, Weissman JS. Cell. 2011 Nov 11;147(4):789-802. doi: 10.1016/j.cell.2011.10.002. Epub 2011 Nov 3. PMID: 22056041; PMCID: PMC3225288.
2) Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. & Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science 324, 218–223 (2009).
3) Kleene, K. C. A possible meiotic function of the peculiar patterns of gene expression in mammalian spermatogenic cells. Mech. Dev. 106, 3–23 (2001).
4) Kleene, K. C. Patterns, mechanisms, and functions of translation regulation in mammalian spermatogenic cells. Cytogenet. Genome Res. 103, 217–224 (2003).
5) Iguchi, N., Tobias, J. W. & Hecht, N. B. Expression profiling reveals meiotic male germ cell mRNAs that are translationally up- and down-regulated. Proc. Natl Acad. Sci. USA 103,7712–7717 (2006).
6) Cardoso-Moreira, M. et al. Gene expression across mammalian organ development. Nature 571, 505–509 (2019).
7) Chen, W. H., Trachana, K., Lercher, M. J. & Bork, P. Younger genes are less likely to be essential than older genes, and duplicates are less likely to be essential than singletons of the same age. Mol. Biol. Evol. 29, 1703–1706 (2012).
Новость подготовили © Финошин А. © Адамейко К.
15.12.2020