
© Симонова Ольга Борисовна, заведующий лабораторией МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИХ ПРОЦЕССОВ РАЗВИТИЯ.
Доктор биологических наук.
Центральная нервная система личинки дрозофилы состоит из 131 077 отдельных клеток. Ученые под руководством профессора университета Флориды (США) Леонида Мороза (к слову сказать, начинавщего свою карьеру в нашем Институте в лаборатории Дмитрия Сахарова) и хорватского нейробиолога из университета в Кембридже (Великобритания) Марты Златич определили транскриптом каждой клетки нервной системы дрозофилы на I, II и III личиночных стадиях развития (1 час, 24 часа и 48 часов после выхода личинки из яйца). Для этого они использовали технику секвенирования РНК одиночных клеток. Дополнительно, для определения пространственного расположения найденных типов клеток в целом мозге они использовали недавно разработанный протокол флуоресцентной РНК-гибридизации in situ (RNA-FISH). В результате впервые был создан полный атлас экспрессии генов центральной нервной системы личинок дрозофилы (Рисунок 1).
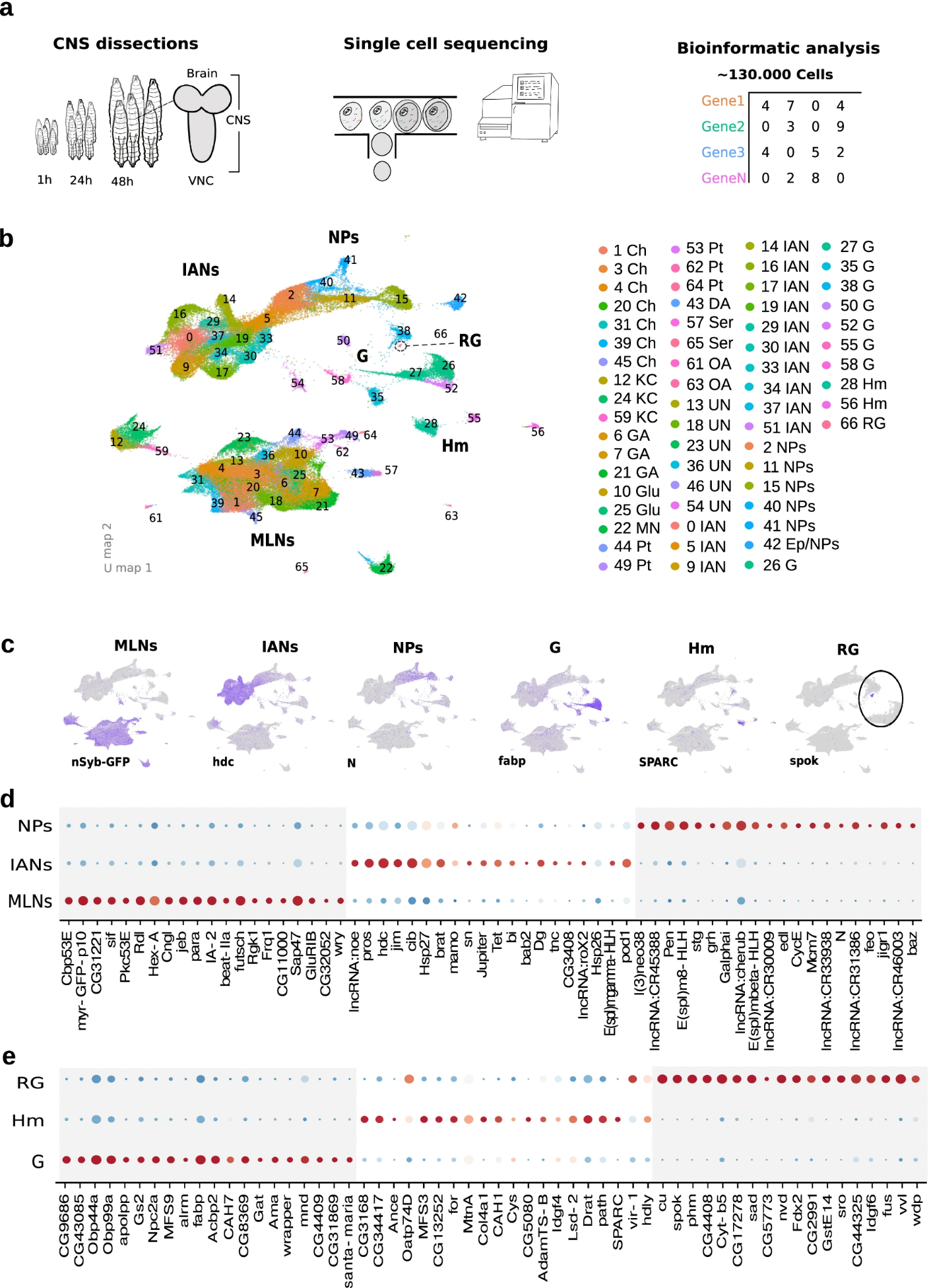
Рис. 1.
Создание атласа экспрессии генов в отдельных клетках нервной системы личинок в возрасте 1, 24 и 48 часов после выхода из яйца. А. Схема эксперимента. Использовали микрожидкостную технологию 10XGenomics для создания библиотеки штрих-кодов отдельных клеток с последующим глубоким секвенированием. Впоследствии использовали Cell Ranger для создания матриц количества клеток по генам и пакет R Seurat для интеграции всех образцов в общее пространство экспрессии генов, позволяющее проводить их совместный анализ. B. 2D-графическое изображение разнообразия типов клеток ЦНС. Каждая точка представляет собой клетку, а их распределение в пространстве зависит от их сходства в профиле экспрессии генов. Каждый кластер имеет цвет и числовой код, как показано в сопроводительной легенде. MLNs: зрелые личиночные нейроны, Ch: холинергические нейроны, KC: клетки Кеньона, GA: гамк-ергические нейроны, Glu: глутаматергические нейроны, MN: моторные нейроны, Pt: пептидергические нейроны, DA: дофаминергические нейроны, Ser: серотонинергические нейроны, OA: октопаминергические нейроны, UN: Неизвестные нейроны, IAN: Незрелые нейроны взрослых, NPs: Нейропредшественники, Ep/NPs: Эпителий/нейропредшественники, G: Глия, Hm: Гемоциты, RG: Кольцевая железа.
На основе изучения паттернов экспрессии генов ученые идентифицировали 67 различных клеточных кластеров (подтипов клеток). Среди них 31 функциональный кластер был обнаружен среди зрелых личиночных нейронов, 1 кластер принадлежал кольцевой железе (орган, отвечающий за гормональное развитие личинки), 8 кластеров принадлежали глиальным клеткам, 6 - предшественникам нейронов (нейробластам) и 13 - развивающимся нейронам будущего взрослого насекомого. Оказалось, что некоторые кластеры присутствуют на всех стадиях развития личинок, в то время как другие зависят от стадии (например, развивающиеся взрослые нейроны).
Созданный Атлас транскриптома клеток нервной системы личинок дрозофилы является ценным ресурсом для биологии развития и системной нейронауки и обеспечивает основу для выяснения того, как гены регулируют развитие и функцию нервной системы.
Известно, что функциональные нейроны различаются по нейротрансмиттерам, которые они выделяют. Такие кластеры и были обнаружены: они содержали холинергические, ГАМКергические, глутаматергические, октопаминергические, серотонинергические, дофаминергические и пептидергические нейроны. Нейроны, участвующие в формировании памяти, и двигательные нейроны, формировали отдельные кластеры, которые сильно отличались с точки зрения экспрессии генов от всех других функциональных личиночных нейронов. Каждый из этих основных классов нейронов избирательно экспрессирует специфический известный маркер (например, гены, участвующие в синтезе или транспорте нейромедиаторов).
Кроме того, были идентифицированы гены с ранее неизвестными функциями, экспрессия которых была сильно обогащена в каждом из этих основных классов. Эти гены являются многообещающими кандидатами в регуляторы функционирования этих классов нейронов. Некоторые из них избирательно экспрессируются только в нейронах одного класса, экспрессия других ограничивается рядом классов с определенными комбинациями трансмиттеров. Информацию об этих генах можно найти в приложениях к статье.
Атлас экспрессии генов на разных стадиях развития личинки предоставляет ресурс для изучения возрастных изменений в личиночных нейронах и спецификации взрослых нейронов.
Учёные отмечают, что новый Атлас также позволит изучить временные изменения в экспрессии генов на разных стадиях развития личинок в определенных типах личиночных нейронов, глиальных клеток, кольцевой железы, развивающихся взрослых нейронов и их клеток-предшественников. Гены, которые по-разному экспрессируются в функциональных личиночных нейронах на поздних стадиях личинки, могут играть роль в регуляции линьки и переходов между стадиями развития, а также в реакции на гормоны, такие как экдизон, ювенильный гормон или проторокальнотропный гормон. Поскольку временные каскады экспрессии факторов транскрипции в нейральных предшественниках определяют судьбу нейронов-потомков, данные, полученные в этом исследовании, предоставляют богатый ресурс для идентификации новых факторов, которые могут быть вовлечены в спецификацию и развитие нейронных судеб.
В заключение хочу отметить, что в марте этого года вышла статья в журнале Science о создании Атласа транскриптома взрослой Drosophila melanogaster: Fly Cell Atlas: A single-nucleus transcriptomic atlas of the adult fruit fly (DOI: 10.1126/science.abk2432). Этот Атлас включает 580 000 клеток и более 250 аннотированных типов клеток (Рисунок 2).
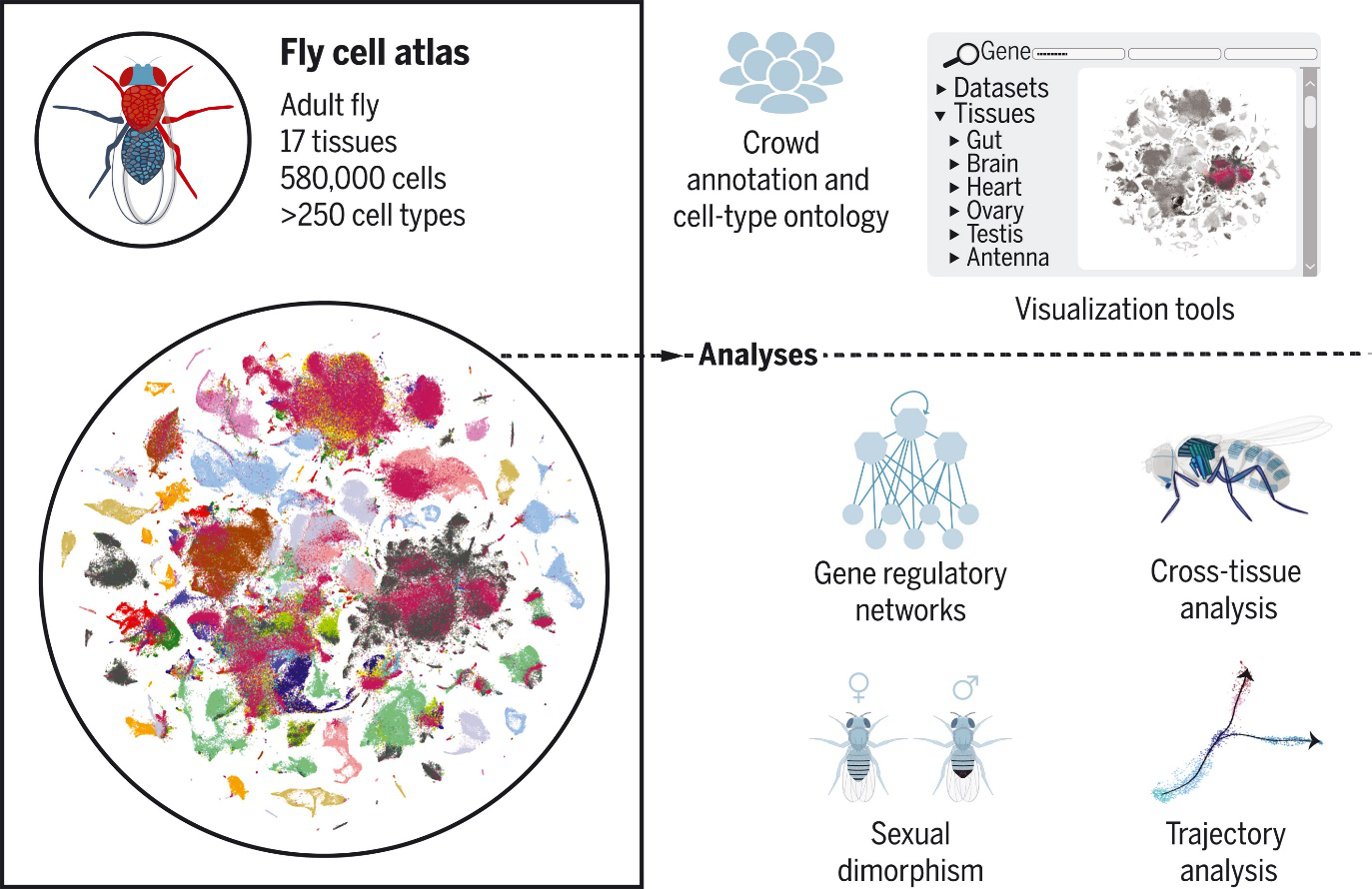
Рис. 2.
Атлас содержит информацию транскриптома индивидуальных клеток (scRNA seq) из 15 тканей самца и самки, а также всей головы и тела. Все данные находятся в свободном доступе для визуализации и загрузки, в правом нижнем углу показаны примеры анализов. Li H, Janssens J, De Waegeneer M, Kolluru SS, Davie K, Gardeux V, Saelens W, David FPA, Brbić M, Spanier K, Leskovec J, McLaughlin CN, Xie Q, Jones RC, Brueckner K, Shim J, Tattikota SG, Schnorrer F, Rust K, Nystul TG, Carvalho-Santos Z, Ribeiro C, Pal S, Mahadevaraju S, Przytycka TM, Allen AM, Goodwin SF, Berry CW, Fuller MT, White-Cooper H, Matunis EL, DiNardo S, Galenza A, O'Brien LE, Dow JAT; FCA Consortium§, Jasper H, Oliver B, Perrimon N, Deplancke B, Quake SR, Luo L, Aerts S, Agarwal D, Ahmed-Braimah Y, Arbeitman M, Ariss MM, Augsburger J, Ayush K, Baker CC, Banisch T, Birker K, Bodmer R, Bolival B, Brantley SE, Brill JA, Brown NC, Buehner NA, Cai XT, Cardoso-Figueiredo R, Casares F, Chang A, Clandinin TR, Crasta S, Desplan C, Detweiler AM, Dhakan DB, Donà E, Engert S, Floc'hlay S, George N, González-Segarra AJ, Groves AK, Gumbin S, Guo Y, Harris DE, Heifetz Y, Holtz SL, Horns F, Hudry B, Hung RJ, Jan YN, Jaszczak JS, Jefferis GSXE, Karkanias J, Karr TL, Katheder NS, Kezos J, Kim AA, Kim SK, Kockel L, Konstantinides N, Kornberg TB, Krause HM, Labott AT, Laturney M, Lehmann R, Leinwand S, Li J, Li JSS, Li K, Li K, Li L, Li T, Litovchenko M, Liu HH, Liu Y, Lu TC, Manning J, Mase A, Matera-Vatnick M, Matias NR, McDonough-Goldstein CE, McGeever A, McLachlan AD, Moreno-Roman P, Neff N, Neville M, Ngo S, Nielsen T, O'Brien CE, Osumi-Sutherland D, Özel MN, Papatheodorou I, Petkovic M, Pilgrim C, Pisco AO, Reisenman C, Sanders EN, Dos Santos G, Scott K, Sherlekar A, Shiu P, Sims D, Sit RV, Slaidina M, Smith HE, Sterne G, Su YH, Sutton D, Tamayo M, Tan M, Tastekin I, Treiber C, Vacek D, Vogler G, Waddell S, Wang W, Wilson RI, Wolfner MF, Wong YE, Xie A, Xu J, Yamamoto S, Yan J, Yao Z, Yoda K, Zhu R, Zinzen RP. Fly Cell Atlas: A single-nucleus transcriptomic atlas of the adult fruit fly. Science. 2022 Mar 4;375(6584):eabk2432. doi: 10.1126/science.abk2432. Epub 2022 Mar 4. PMID: 35239393; PMCID: PMC8944923.
Это мега исследование представляет собой совокупный труд 43 ученых из США, Европы и Азии (Корея) и финансировалось 20 грантами. Углубленный анализ выявил редкие типы клеток, специфические для клеточного типа генные сигнатуры и половой диморфизм. Такой Атлас предоставляет научному сообществу ресурс для изучения генетических нарушений и болезней с разрешением в одну клетку.
Новость представила ©Симонова О.Б.
05.09.2022